U-Net을 망막 혈관 세그멘테이션(retinal blood vessel segmentation) 문제에 적용해 보자.
아래 사이트에 가면 데이터셋과 관련 논문, 그리고 텐서플로나 파이토치로 작성한 코드들이 많이 나온다.
Papers with Code - Retinal Vessel Segmentation
Retinal vessel segmentation is the task of segmenting vessels in retina imagery. <span style="color:grey; opacity: 0.6">( Image credit: [LadderNet](https://github.com/juntang-zhuang/LadderNet) )</span>
paperswithcode.com
사이트에는 4가지 데이터셋이 나와 있는데, 이중 DRIVE (Digital Retinal Images for Vessel Extraction)을 사용한다. DRIVE 데이터셋은 아래 사이트에서도 구할 수 있다.
DRIVE Dataset - Hub | Activeloop
Paper: Staal, J., Abràmoff, M. D., Niemeijer, M., Viergever, M. A., & Van Ginneken, B. (2004). Ridge-based vessel segmentation in color images of the retina. IEEE transactions on medical imaging, 23(4), 501-509.
docs.activeloop.ai
DRIVE 데이터셋은 학습용 셋20개와 테스트용 셋 20개가 들어있다. 각 데이터셋은 512x512 망막 이미지와 각 망막 이미지의 모든 픽셀에 대해서 그 픽셀이 혈관에 속하는지 아닌지 수동으로 라벨링한 세그멘테이션 이미지로 구성되어 있다.

데이터셋이 겨우 20개 밖에 안되므로 U-Net 학습에 앞서서 우선 데이터셋을 늘릴 필요가 있다. 이미지를 회전하거나, 수평 또는 수직으로 뒤집는 방법으로 데이터 수를 늘릴 수 있다. 이를 데이터 증강(data augmentation)이라고 한다. 다음 사이트에 가면 다양한 방법과 예제 코드가 게시되어 있다.
데이터 증강 | TensorFlow Core
5월 11~12일 Google I/O에서 TensorFlow에 참여하세요. 지금 등록하세요. 데이터 증강 개요 이 튜토리얼에서는 이미지 회전과 같은 무작위(그러나 사실적인) 변환을 적용하여 훈련 세트의 다양성을 증가
www.tensorflow.org
데이터 증강 방법을 통하여 학습용 데이터셋을 20개에서 80개로 늘린다. 그리고 학습용 데이터셋의 망막 이미지와 세그멘테이션 이미지를 각각 train/image와 train/mask 폴더에 저장하고 테스트용 데이터셋도 각각 test/image와 test/mask 폴더에 저장한다. 망막 이미지 파일의 이름과 해당 세그멘테이션 이미지 파일의 이름을 동일하게 만드는 게 편리하다.

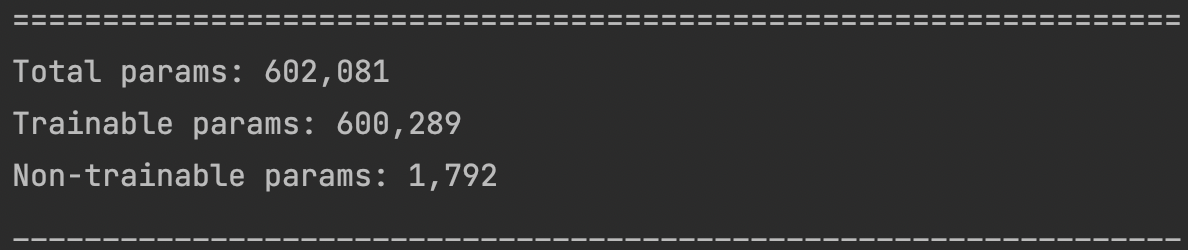
원래 U-Net 모델에서 사용된 컨볼루션 필터의 개수는 인코딩과 디코딩의 각 단계에서 64, 128, 256, 512개이고 브릿지에서는 1024개인데, 그러면 학습해야 할 파라미터 수가 3,100만개에 달한다.

노트북의 성능에 비해 지나치게 많으므로 컨볼루션 필터의 수를 16, 32, 48, 64, 128 로 줄인다.
# Encoder (original 64, 128, 256, 512)
self.e1 = EncoderBlock(16)
self.e2 = EncoderBlock(32)
self.e3 = EncoderBlock(48)
self.e4 = EncoderBlock(64)
# Bridge (original 1024)
self.b = ConvBlock(128)
# Decoder (original 512, 256, 128, 64)
self.d1 = DecoderBlock(64)
self.d2 = DecoderBlock(48)
self.d3 = DecoderBlock(32)
self.d4 = DecoderBlock(16)
그러면 학습해야 할 파라미터 수는 60만개 정도로 준다.
전에 만들어 논 U-Net 모델을 컴파일 한다. 세그멘테이션은 혈관인지 아닌지만 구별하면 되므로 클래스 수는 1개로 하고 손실함수는 이진 크로스엔트로피(binary cross entroy) 함수를 사용한다. 카테고리가 여러 개인 경우를 위해서 몇가지 손실함수가 개발되어 있으나 이에 대해서는 다음에 알아보기로 한다.
# create unet
n_classes = 1
self.unet = UNET(n_classes)
# compile
lr = 1e-4
self.unet.compile(
loss=tf.keras.losses.BinaryCrossentropy(),
optimizer=tf.keras.optimizers.Adam(lr),
metrics=['accuracy']
)
학습은 model.fit 함수를 이용한다.
(x_train, y_train), (_, _) = get_data()
if os.path.exists('./save_weights/unet.h5'):
self.unet.load_weights("./save_weights/unet.h5")
history = self.unet.fit(
x_train, y_train, epochs=epochs, batch_size=4,
shuffle=True, validation_split=0.2, verbose=2)
에폭을 100으로 했을 때 학습 곡선은 다음과 같다.

다음은 테스트 셋을 이용하여 혈관을 제대로 검출했는지 그림으로 비교한 것이다.
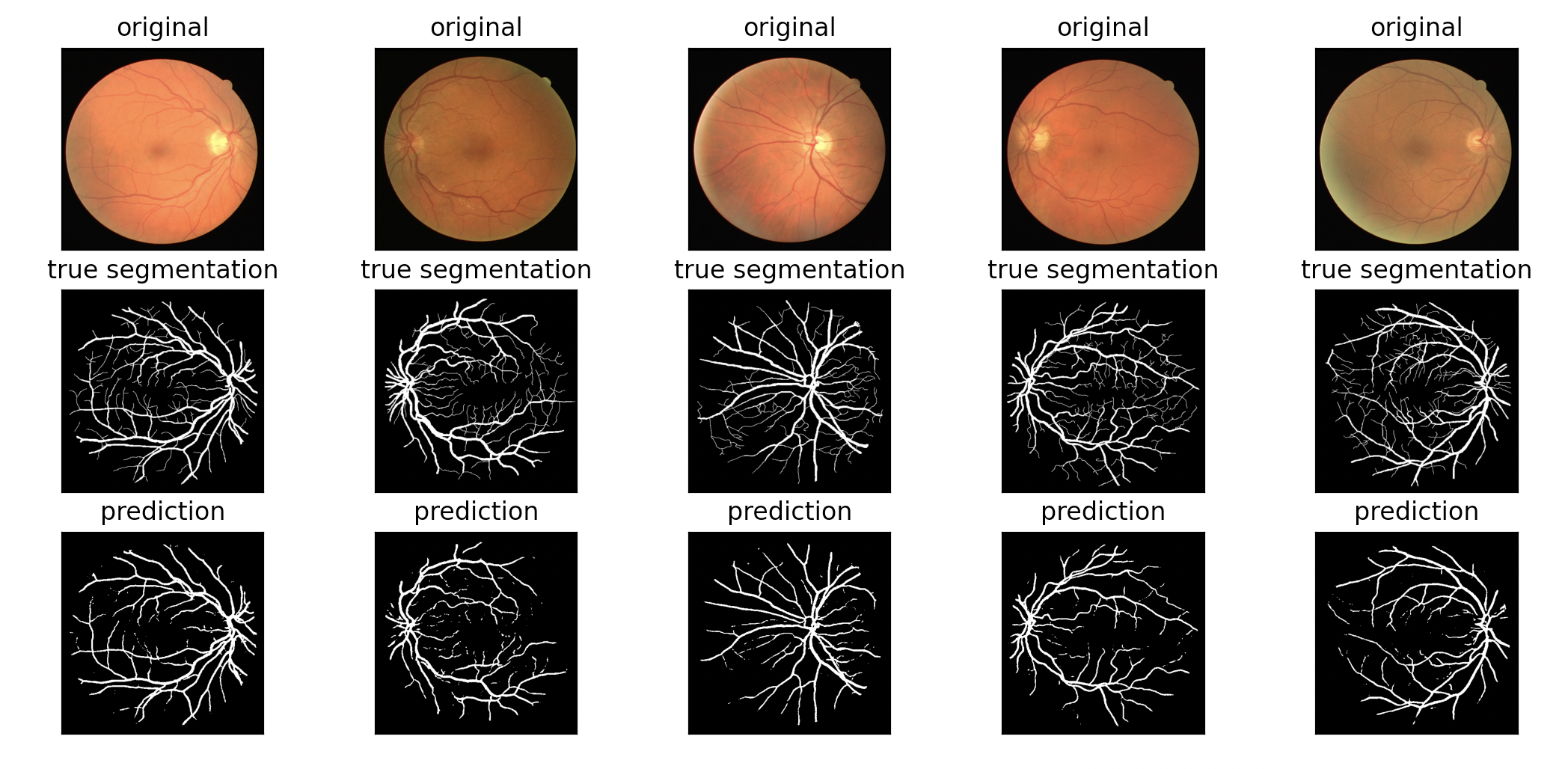
가운데 그림은 참값, 맨 아래 그림은 예측값이다. 미세한 혈관까지는 파악하지 못했으나 적은 데이터에도 불구하고 전체적으로 만족스러운 성능을 보였다.
그림 그릴 때, OpenCV는 BGR 포맷을 사용하므로 matplotlib 패키지를 이용할 때는 RGB로 바꿔줘야 한다는 점에 주의해야 한다.
image = cv2.cvtColor(pred, cv2.COLOR_BGR2RGB)
plt.imshow(image)
U-Net을 학습하기 위한 전체 코드는 다음과 같다.
unet_train.py
import os
import random
from unet_model import UNET
from data import Data
import tensorflow as tf
import cv2
import matplotlib.pyplot as plt
""" loading training and test data """
def get_data():
retina = Data('aug_data/')
return retina.load_data()
""" setup unet agent """
class UNETagent(object):
def __init__(self):
# create unet
n_classes = 1
self.unet = UNET(n_classes)
# compile
lr = 1e-4
self.unet.compile(
loss=tf.keras.losses.BinaryCrossentropy(),
optimizer=tf.keras.optimizers.Adam(lr),
metrics=['accuracy']
)
#self.unet.build(input_shape=(None, 512, 512, 3)) # (batch, H, W, C)
#self.unet.summary()
""" evaluation """
def eval(self, x_test, y_test):
# evaluate
if os.path.exists('./save_weights/unet.h5'):
self.unet.load_weights("./save_weights/unet.h5")
else:
return 0
pred_seg = self.unet(x_test)
plt.figure(figsize=(20, 6))
n = 5
ini = random.randint(0, 20-n)
for i in range(n):
# display image
ax = plt.subplot(3, n, i + 1)
image = cv2.cvtColor(x_test[ini+i].numpy(), cv2.COLOR_BGR2RGB)
plt.imshow(image)
plt.title("original")
ax.get_xaxis().set_visible(False)
ax.get_yaxis().set_visible(False)
# display true segmentation
ax = plt.subplot(3, n, i + 1 + n)
image = cv2.cvtColor(y_test[ini+i].numpy(), cv2.COLOR_BGR2RGB)
plt.imshow(image)
plt.title("true segmentation")
ax.get_xaxis().set_visible(False)
ax.get_yaxis().set_visible(False)
# display predictions
ax = plt.subplot(3, n, i + 1 + n + n)
pred = pred_seg[ini + i].numpy()
pred[pred > 0.5] = 1
pred[pred <= 0.5] = 0
image = cv2.cvtColor(pred, cv2.COLOR_BGR2RGB)
plt.imshow(image)
plt.title("prediction")
ax.get_xaxis().set_visible(False)
ax.get_yaxis().set_visible(False)
plt.show()
""" training """
def train(self, epochs):
(x_train, y_train), (_, _) = get_data()
if os.path.exists('./save_weights/unet.h5'):
self.unet.load_weights("./save_weights/unet.h5")
history = self.unet.fit(
x_train, y_train, epochs=epochs, batch_size=4,
shuffle=True, validation_split=0.2, verbose=2)
plt.figure(figsize=(10, 4))
plt.subplot(1, 2, 1)
plt.plot(history.history['loss'], 'b-', label='loss')
plt.plot(history.history['val_loss'], 'r-', label='val_loss')
plt.xlabel('epoch')
plt.legend()
plt.subplot(1, 2, 2)
plt.plot(history.history['accuracy'], 'g-', label='accuracy')
plt.plot(history.history['val_accuracy'], 'k-', label='val_accuracy')
plt.xlabel('epoch')
plt.legend()
plt.show()
# save
self.unet.save_weights("./save_weights/unet.h5")
if __name__ == "__main__":
agent = UNETagent()
agent.train(100)
'AI 딥러닝 > DLA' 카테고리의 다른 글
| [VAE] beta-VAE (0) | 2023.05.11 |
|---|---|
| [VAE] 변이형 오토인코더(Variational Autoencoder) (0) | 2023.04.30 |
| [U-Net] U-Net 구조 (0) | 2022.05.11 |
| [PINN] 버거스 방정식 기반 신경망 (Burgers’ Equation-Informed Neural Network) 코드 업데이트 (0) | 2022.01.11 |
| [PINN] 비압축성 유체 정보 기반 신경망 (Incompressible NS-Informed Neural Network) (0) | 2021.11.02 |




댓글